O diagnóstico molecular é uma ferramenta indispensável na saúde pública atual, especialmente no diagnóstico de infecções causadas por vírus de RNA. A técnica de PCR, quando associada à etapa de transcrição reversa (RT), permite identificar com precisão o material genético viral em amostras clínicas. A compreensão desses procedimentos laboratoriais e de seus fundamentos biotecnológicos é relevante no ENEM, já que envolve a integração dos conhecimentos de biologia molecular e genética em contextos reais.
Entre as diversas técnicas para diagnóstico da covid-19, destaca-se o teste genético. Considerando as diferentes variantes e cargas virais, um exemplo é a PCR, reação efetuada por uma enzima do tipo polimerase.
Essa técnica permite identificar, com confiabilidade, o material genético do SARS-CoV-2, um vírus de RNA. Para comprovação da infecção por esse coronavírus, são coletadas amostras de secreções do indivíduo. Uma etapa que antecede a reação de PCR precisa ser realizada para permitir a amplificação do material genético do vírus.
Essa etapa deve ser realizada para
A) concentrar o RNA viral para otimizar a técnica.
B) identificar nas amostras anticorpos anti-SARS-CoV-2.
C) proliferar o vírus em culturas, aumentando a carga viral.
D) purificar ácidos nucleicos virais, facilitando a ação da enzima.
E) obter moléculas de cDNA viral por meio da transcrição reversa.
A Reação em Cadeia da Polimerase (Polymerase Chain Reaction ou PCR) é uma técnica muito utilizada em estudos biologia molecular, bem como para análises clínicas.
O objetivo primário da PCR é a amplificação de fragmentos específicos de DNA, obtidos a partir de uma amostra. Partindo de um número reduzido de fragmentos de DNA – cuja quantidade não permitiria sua utilização em estudos, testes e diagnósticos – a técnica de PCR possibilita a geração de grande número de cópias dessas moléculas, ou seja sua amplificação. A amplificação gera quantidade suficiente dos fragmentos desejados para utilização em laboratório.
Para compreendermos a PCR, é necessário analisar a estrutura do DNA e seu mecanismo de replicação.
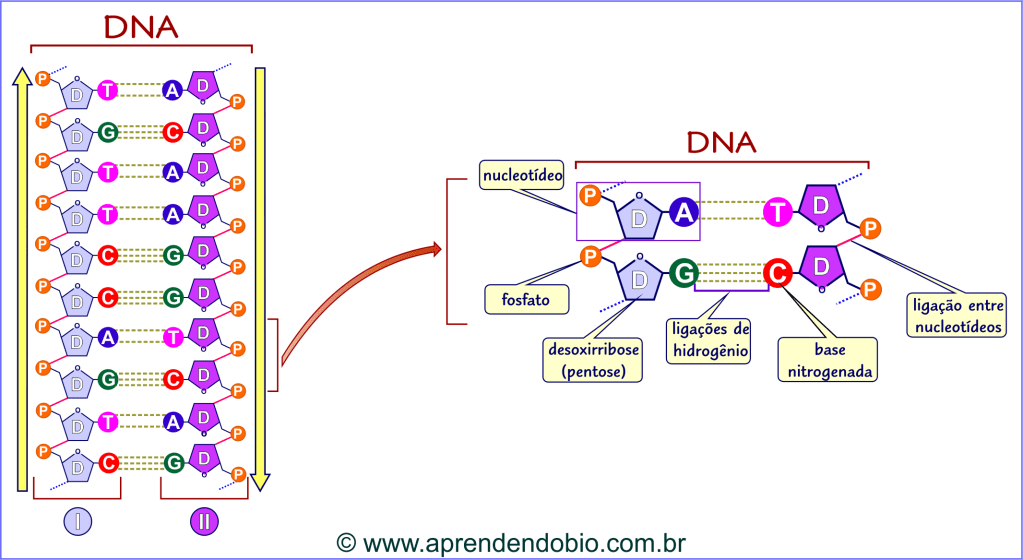
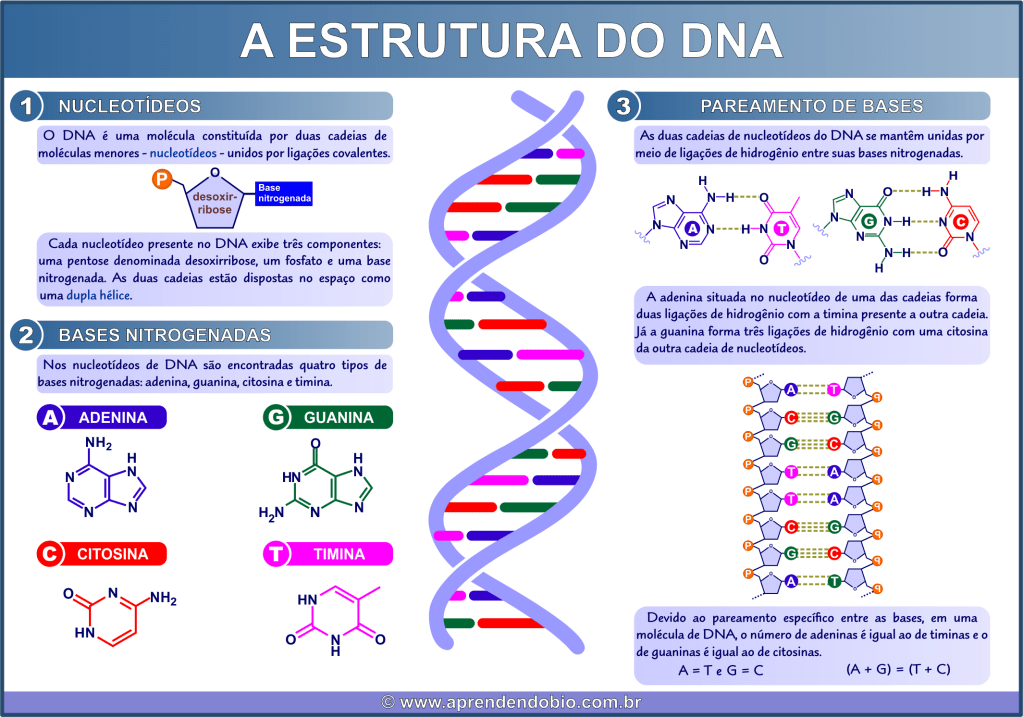
O ácido desoxirribonucleico ou DNA é uma molécula grande (isto é, uma macromolécula) resultante da ligação entre inúmeras moléculas menores denominadas nucleotídeos. Cada nucleotídeo, por sua vez, é formado por três componentes:
– desoxirribose: uma pentose, ou seja, um açúcar (carboidrato) com cinco carbonos.
– um resíduo de fosfato (PO4-3)
– uma base nitrogenada, que pode ser adenina (A), guanina (G), citosina (C) ou timina (T).
A figura a seguir ilustra, de forma simplificada, a estrutura de um nucleotídeo.
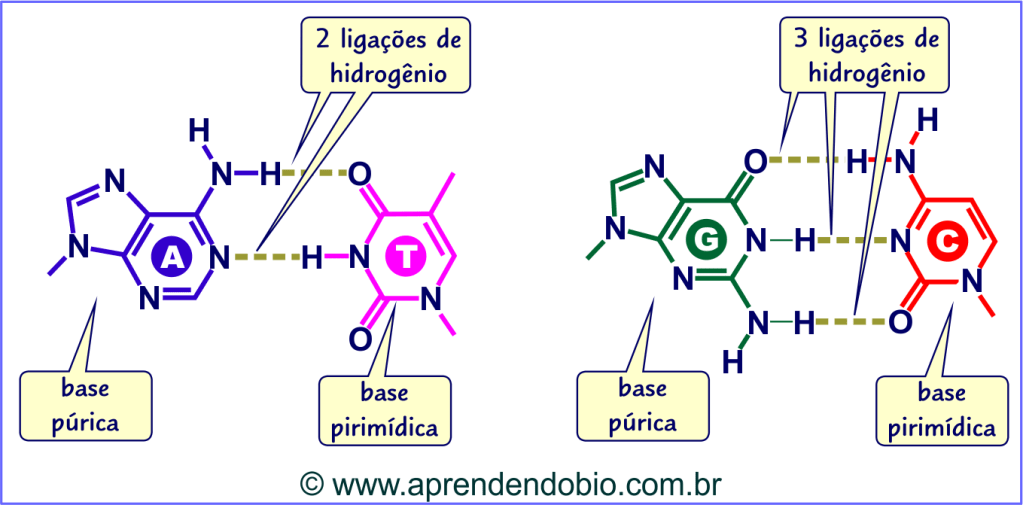
As bases nitrogenadas são moléculas com estrutura em cadeia fechada (cíclica). Dependendo do número de cadeias cíclicas (“anéis”) presentes na base, ela poderá ser classificada como purina (base púrica) ou pirimidina (base pirimídica). As moléculas de adenina e guanina exibem, cada uma delas, estrutura com duas cadeias cíclicas e, por isso, são classificadas como purinas. Já citosina e timina, cujas moléculas apresentam uma única cadeia cíclica, são pirimidinas.
Nas células, a molécula de DNA é formada por duas cadeias de nucleotídeos (vírus podem apresentar DNA com uma única cadeia). Essas cadeias se mantêm unidas por interações intermoleculares, do tipo ligação de hidrogênio, estabelecidas entre suas bases nitrogenadas. Para isso há um pareamento específico entre as bases das duas cadeias: a adenina de uma cadeia forma duas ligações de hidrogênio com a timina da outra cadeia, enquanto a guanina situada em uma das cadeias estabelece três ligações de hidrogênio com a citosina da outra cadeia.
Observe que o pareamento para formação das ligações de hidrogênio se dá entre uma base púrica e uma pirimídica: a púrica adenina com a pirimídica timina e a púrica guanina com a pirimídica citosina.
Os nucleotídeos que formam cada cadeia estão covalentemente ligados. Essa ligação é estabelecida entre a desoxirribose (pentose) de um nucleotídeo e o fosfato do nucleotídeo vizinho – na mesma cadeia.
Essas cadeias têm orientação oposta (invertida) uma em relação à outra e, por isso, são antiparalelas. Na figura a seguir são apresentados alguns detalhes da estrutura molecular do DNA, dentre os quais a orientação antiparalela (setas amarelas) das cadeias I e II.
Na realidade, as cadeias antiparalelas que formam a molécula de DNA estão dispostas no espaço como espirais, constituindo o que se convencionou chamar de dupla-hélice. A dupla-hélice se assemelha a uma escada em espiral, na qual os corrimãos são representados pela ligação entre as desoxirriboses e fosfatos dos nucleotídeos de cada cadeia. Já os degraus são os pares de bases nitrogenadas unidas pelas ligações de hidrogênio.
Devido ao pareamento específico entre as bases (A-T e G-C), podemos concluir que, em uma mesma molécula de DNA, o número de bases nitrogenadas adenina é igual ao número de timinas, assim como o número de guaninas é igual ao de citosinas.
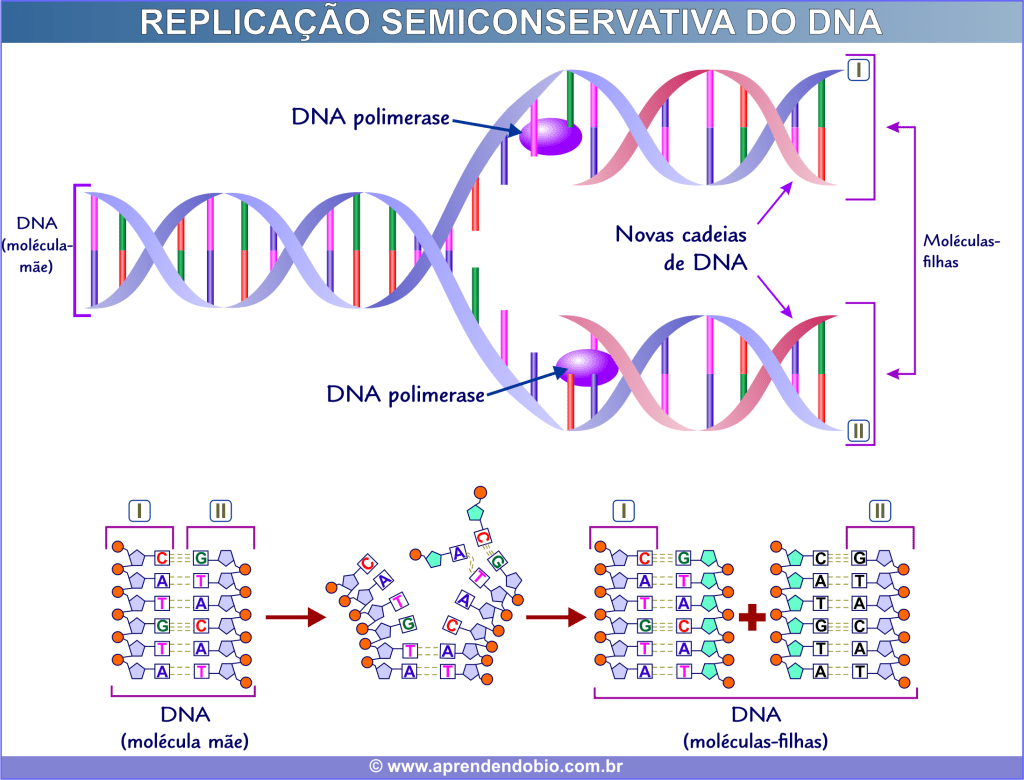
As células possuem um mecanismo que as possibilita produzir cópias exatas de suas moléculas de DNA. Antes de se dividir, é necessário duplicar as moléculas de DNA para distribuí-las igualmente às células-filhas.
Esse processo – denominado replicação – envolve um conjunto de enzimas, dentre as quais se destaca a DNA-polimerase. Durante a replicação, as duas cadeias são separadas, ou seja, as ligações de hidrogênio que as mantêm são desfeitas.
A partir daí, a DNA-polimerase posiciona nucleotídeos livres em uma sequência determinada pelas bases de cada cadeia original. Sendo assim, são produzidas duas novas cadeias de nucleotídeos cujas sequências de bases nitrogenadas são complementares ao de cada cadeia original. À medida que a nova cadeia é produzida (= polimerizada), formam-se as ligações de hidrogênio entre as bases da cadeia original e aquelas da nova cadeia.
Como resultado são produzidas duas novas moléculas de DNA (cada uma delas com duas cadeias de nucleotídeos) com sequências de bases exatamente iguais.
A replicação do DNA é semiconservativa, uma vez que cada uma das novas moléculas (moléculas-filhas) fica com uma das cadeias da molécula original (molécula-mãe), como é possível verificar nas representações a seguir.
A amplificação de fragmentos de DNA por meio da PCR envolve um procedimento baseado na replicação realizada pelas células. Os fragmentos que se deseja amplificar (replicar) são colocados, juntamente com nucleotídeos livres e outros reagentes no interior de um equipamento denominado termociclador.
O nome do equipamento deriva do fato de que, diferentemente do que ocorre nas células, o processo de amplificação envolve variações na temperatura do meio. Incialmente, aumenta-se a temperatura para possibilitar a separação das cadeias de DNA. Em seguida, a temperatura é reduzida de forma que a enzima DNA-polimerase (um dos reagentes adicionados) possa produzir as cadeias complementares e para que as ligações de hidrogênio se formem entre os pares de bases.
A DNA-polimerase utilizada na PCR é conhecida como taq-polimerase. Esta enzima foi obtida a partir da bactéria Thermus aquaticus, que habita fontes hidrotermais (fontes de água quente). A taq-polimerase é capaz de realizar sua função sem que sua estrutura seja afetada pelas temperaturas elevadas utilizadas durante o processo de amplificação.
Para que a enzima taq-polimerase possa atuar sobre o fragmento de DNA que se deseja multiplicar (amplificar) é necessária uma pequena cadeia simples, também de DNA, conhecida como primer (iniciador). Esta cadeia emparelha-se, por complementaridade de bases, ao fragmento de DNA a ser amplificado e serve como ponto de início para a ação replicadora da taq-polimerase.
A técnica da PCR segue os passos descritos a seguir.
1. A solução contendo os fragmentos de DNA que serão amplificados é colocada no termociclador juntamente com os primers, com a enzima taq-polimerase, Mg+2 (cofator necessário à atividade da taq-polimerase) e com os nucleotídeos livres.
2. Aumenta-se a temperatura da solução para, aproximadamente, 94°C. Isto faz com que as duas cadeias do DNA se separem, uma vez que a temperatura elevada desfaz as ligações de hidrogênio entre as bases nitrogenadas. Essa etapa se denomina desnaturação.
3. A seguir, a temperatura é reduzida para 50°C, permitindo que os primers se pareiem às cadeias previamente separadas. Essa etapa de pareamento dos primers com os fragmentos de DNA recebe o nome de anelamento.
4. Após o anelamento, tem início a etapa de extensão ou polimerização, na qual a temperatura da solução contida no termociclador é ajustada para 72°C, ideal para a taq-polimerase, que entra em ação replicando os fragmentos de DNA.
O ciclo, constituído pelas três etapas (desnaturação, anelamento e extensão), é repetido várias vezes. Após cada ciclo, o número de cópias do DNA é duplicado em relação ao anterior.
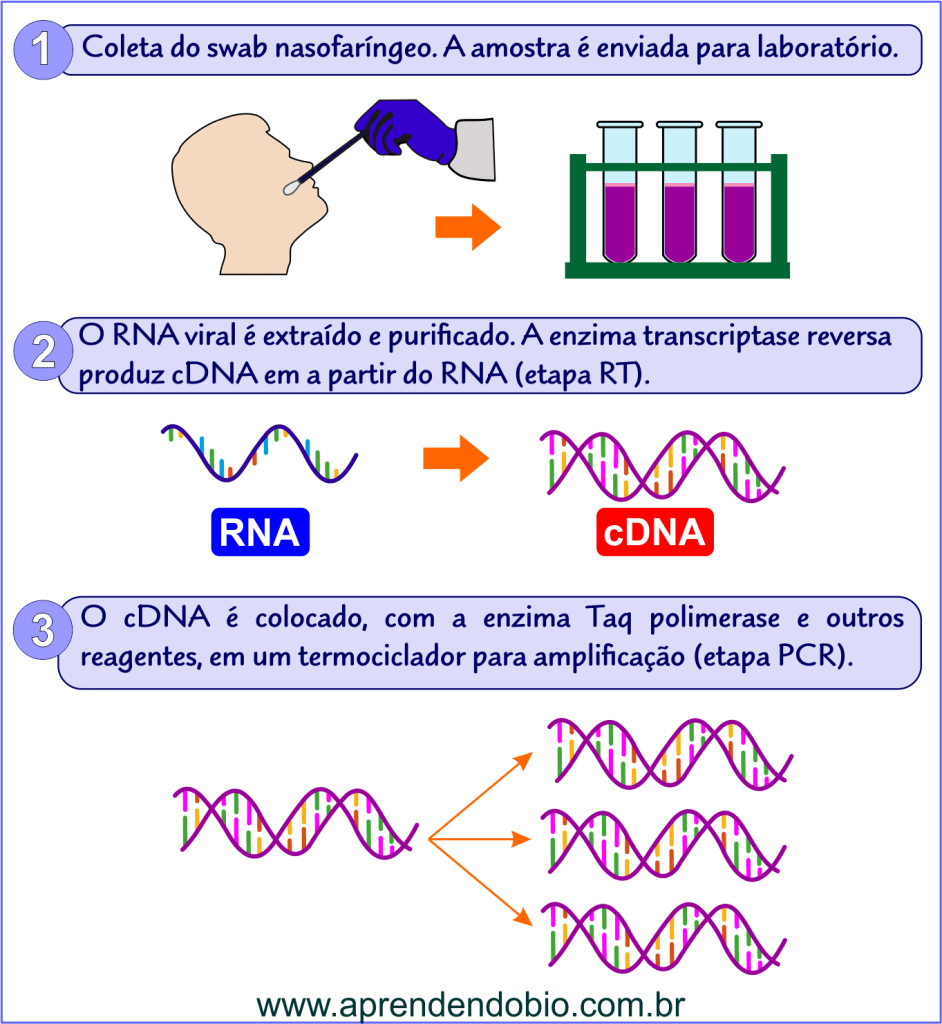
A PCR é útil para o diagnóstico da COVID-19 pois permite a amplificação de possível material genético do SARS-Cov-2 a partir das amostras de secreções nasais ou da orofaringe obtidas por meio do swab (uma espécie de “cotonete”).
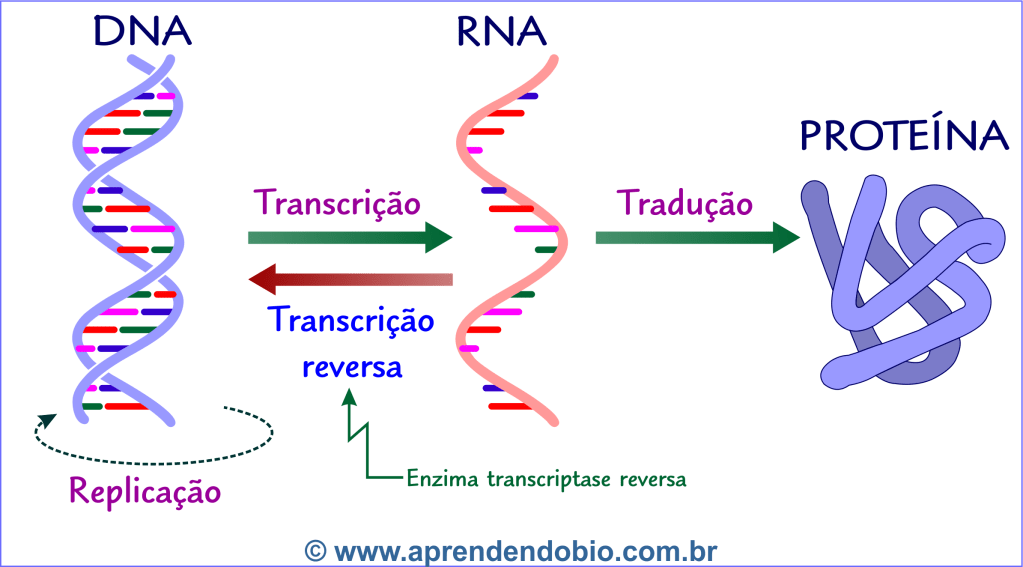
Como a PCR é um procedimento para amplificar fragmentos de DNA e o SARS-Cov-2 é um vírus cujo genoma é de RNA, é necessário, antes dos de iniciar os ciclos de amplificação, obter uma molécula de DNA, a partir do RNA viral. Isso é possível com a utilização da enzima transcriptase reversa.
O RNA, assim como o DNA, é um ácido nucleico e, por isso, constituído por nucleotídeos. Entretanto, os nucleotídeos do RNA apresentam a pentose denominada ribose e suas bases nitrogenadas são adenina (A), guanina (G), citosina (C) e uracil (U). Além disso, o RNA exibe apenas uma cadeia de nucleotídeos.
As enzimas encontradas nas células são capazes de gerar moléculas de RNA utilizando uma das cadeias de DNA como molde. Esse processo se chama transcrição. A informação genética contida no RNA transcrito é, então, traduzida em uma sequência de aminoácidos de uma proteína.
Para utilizar a PCR no diagnóstico da COVID-19 é necessário realizar uma transcrição reversa (reverse transcription ou RT). É nesse momento que a enzima transcriptase reversa se mostra útil.
Essa enzima, presente em certos vírus – como o HIV e o vírus da hepatite A –, é capaz de gerar uma molécula de DNA a partir de uma cadeia molde de RNA, ou seja, realizar a transcrição reversa. A molécula de DNA resultante da atividade da enzima transcriptase reversa é denominada cDNA (complementary DNA).
Se a amostra colhida pelo swab contiver o SARS-Cov-2, o RNA desse vírus será utilizado na transcrição reversa (etapa RT) para obtenção do cDNA. Em seguida, o cDNA é amplificado por meio de vários ciclos de PCR (desnaturação, anelamento e extensão) gerando quantidades que podem ser detectadas para diagnóstico da COVID-19. O procedimento descrito, transcrição reversa (RT) seguida de amplificação pela reação em cadeia da polimerase (PCR), recebe a denominação de RT-PCR e constitui o exame indicado para diagnóstico da COVID-19.
O exame de RT-PCR é considerado o mais adequado (padrão-ouro) para diagnóstico da COVID-19. A PCR é um procedimento da biologia molecular cujo objetivo é produzir grande número cópias de fragmentos específicos de DNA (amplificação do DNA).
Para realização da PCR, os fragmentos de DNA a serem amplificados são adicionados, juntamente com a enzima taq-polimerase e outros reagentes (como nucleotídeos livre e íons magnésio), em um equipamento denominado termociclador.
Nesse equipamento ocorre a replicação dos fragmentos de DNA pela enzima taq polimerase em um processo denominado reação em cadeia da polimerase (PCR). A taq-polimerase utiliza os nucleotídeos livres para produzir cópias dos fragmentos de DNA, aumentando (amplificando) a concentração dessas moléculas.
O vírus SARS-Cov-2, causador da COVID-19 tem genoma de RNA. Então, para que a PCR seja utilizada no diagnóstico dessa enfermidade, é necessária a realização de uma etapa na qual o RNA viral é utilizado como molde para gerar uma molécula de DNA (cDNA). Essa etapa é denominada transcrição reversa (etapa RT).
Uma vez obtido o cDNA, essa molécula é multiplicada (amplificada) por meio do processo de PCR. Esse procedimento – transcrição reversa (etapa RT) seguida da PCR – é conhecida como RT-PCR, o exame utilizado para diagnóstico da COVID-19.
Sendo assim, a etapa RT, que antecede a reação de PCR, precisa ser realizada para permitir a amplificação do material genético do vírus, pois possibilita obter moléculas de cDNA viral por meio da transcrição reversa.
🧬 Sustente o conhecimento que transforma
Se esta explicação ajudou você a compreender como a RT-PCR permite identificar o SARS-CoV-2 com precisão molecular, considere apoiar o Aprendendo Biologia.
Gestos simples — compartilhar, divulgar ou contribuir financeiramente — sustentam a produção independente de conteúdo, fortalecem a rede de aprendizagem e ampliam o alcance da educação em biologia.
📩 Chave Pix: contato@aprendendobio.com.br

Obrigado por apoiar a educação científica com propósito!
Saiba mais